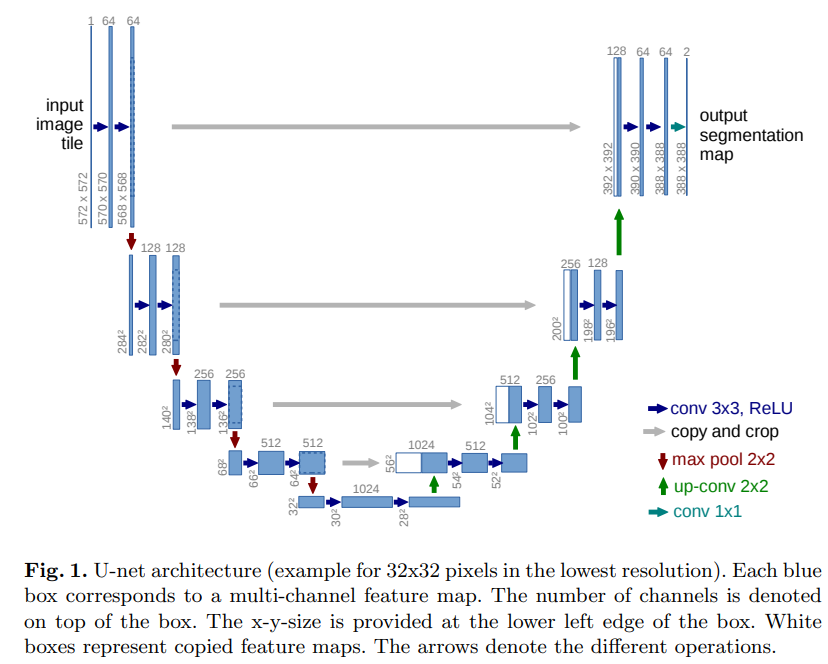
이번엔 기말고사 전에 리뷰했던 U-Net을 구현해보는 시간을 가져볼 것이다. 리뷰했을 때에 언급했지만 U-Net은 biomedical dataset에 특화된 모델이어서 sub task 역시 bio에 관련된 것으로 가져왔다.
1. Task와 Dataset 소개
오늘의 dataset은 아래에 올라와있다.
https://www.kaggle.com/datasets/andrewmvd/cancer-inst-segmentation-and-classification
Cancer Instance Segmentation and Classification 1
(Part 1/3) 200k labeled nuclei of 19 tissue types
www.kaggle.com
전체 데이터셋은 크게 3개로 나눠있는데 하나의 part만 가져와도 256*256의 이미지가 2656개가 있어서 충분하다고 판단해(사실 충분한단 것은 gpu memory의 한계를 의미하는 것과 같다.) part 1만 사용하였다.
Author Notes의 README를 읽어보면 19개의 tissue types의 데이터로 이루어져있으며 각각은 0: Neoplastic cells, 1: Inflammatory, 2: Connective/Soft tissue cells, 3: Dead Cells, 4: Epithelial, 6: Background 이렇게 마스킹 되어있다.
용어를 하나하나 살펴보면
Neoplastic cells : 종양을 형성하는 세포
Inflammatory : 염증성
Connective/Soft tissue cells: 결합조직 및 연조직으로 신체의 구조와 지지 기능을 담당하는 세포들
Dead Cells: 죽은 세포, 더 이상 기능하지 않는 세포
Epithelial: 상피 세포, 신체의 표면과 내부 장기를 덮고 있는 세포
인데 오랜만에 생물 공부를 하는 느낌이다.
그리고 추가적으로 데이터 형식을 살펴보면 특이하게 images와 masks가 images.npy, masks.npy로 되어 있다는 것이다.
(npy 파일은 NumPy 라이브러리에서 사용하는 파일 형식으로, 다차원 배열 데이터를 효울적으로 저장하고 로드하는데 사용된다.)
import numpy as np
images = np.load(r'C:\Users\james\Desktop\U-net\Part 1\Part 1\Images\images.npy', mmap_mode='r')
images = images.astype('int32')
masks = np.load(r'C:\Users\james\Desktop\U-net\Part 1\Part 1\Masks\masks.npy', mmap_mode='r')
masks = masks.astype('int32')
print(images.shape)
print(masks.shape)(코드의 mmap_mode 매개변수는 매모리 맵핑을 사용하여 대용량 배열 데이터를 디스크에서 직접 읽는 방식을 지정하는데 사용한다. 이 값을 기본값으로하게 되면 매모리 맵핑을 사용하지 않아 파일 전체가 메모리에 로드되는데 파일 양이 커서 local의 경우 과부화가 걸린다. 따라서 읽기 전용 모드로 파일을 메모리에 매핑하여 데이터의 일부만 로드하고 필요한 부분만 읽게 하였다. 즉, 메모리 사용량을 줄이고 입출력 성능을 향상시키기 위해 하는 것!)
(2656, 256, 256, 3)
(2656, 256, 256, 6) 출력해보면 masks의 채널이 왜 6인지는 위에 데이터셋을 보면 알 수 있을 것이다. 일전의 FCN에서 했던 cloth segmentation의 경우 semantic segmentation이기 때문에 2차원 mask에서 각 pixel의 값에 class의 number를 매핑하면 충분하였다. 하지만 이번 task는 instance segmentation을 해야하기 때문에 3차원 mask에 각 채널이 하나의 class를 의미하고 각 class에 해당하는 instance를 숫자로 매핑하게 되었다.
Semantic segmentation과 instance segmentation이 무엇이지 까먹었다면 이 곳으로
[Paper Review] Fully Convolutional Netowrks for Semantic Segmentation
티스토리의 첫 번째 포스트는 Jonathan Long, Evan Shelhamer, Trevor Darrell의 논문인 Fully Convolutional Networks for Semantic Segmentation에 대해 리뷰해 보겠다. 기존에 velog에 있지만 카테고리 정리에 유리한 tistory
go-big-or-go-home.tistory.com
그럼 task와 데이터 소개를 마쳤으니 본격적인 구현 단계로 넘어가 보겠다.
2. Dataset 정의 및 Dataset 로딩
import torch
from torch.utils.data import Dataset, DataLoader
from PIL import Image
class NumpySegDataset(Dataset):
def __init__(self, images_path, masks_path, transform=None, target_transform=None):
self.images = np.load(images_path, mmap_mode='r')
self.masks = np.load(masks_path, mmap_mode='r')
self.transform = transform
self.target_transform = target_transform
def __len__(self):
return len(self.images)
def __getitem__(self, idx):
image = self.images[idx]
mask = self.masks[idx]
if self.transform:
image = self.transform(image)
if self.target_transform:
mask = self.target_transform(mask)
# image는 정규화하면 소수로 변하니까 float, mask는 정규화 안하니까 int
image = torch.tensor(image, dtype=torch.float32).permute(2,0,1)
mask = torch.tensor(mask, dtype=torch.int64).permute(2,0,1)
return image, mask항상 해왔듯이 Dataset class인 NumpySegDataset class를 정의하고 __init__,. __len__함수와 __getitem__ 함수를 정의하였다. 다만 저번 미니 프로젝트와는 다른 점은 __init__ 함수를 만들 때 numpy 배열이기 때문에 np.load와 mmap_mode = 'r'이라는 점이다.
images_path=r'C:\Users\james\Desktop\U-net\Part 1\Part 1\Images\images.npy'
masks_path=r'C:\Users\james\Desktop\U-net\Part 1\Part 1\Masks\masks.npy'
dataset = NumpySegDataset(images_path, masks_path)이렇게 dataset을 정의해주었고
total_len = len(dataset)
train_len = int(total_len * 0.8)
val_len = total_len - train_len
train_dataset, val_dataset = torch.utils.data.random_split(dataset, [train_len, val_len])
train_loader = DataLoader(train_dataset, batch_size=4, shuffle=True)
val_loader = DataLoader(val_dataset, batch_size=4, shuffle=True) dataset을 로딩해줄 때 8:2의 비율로 train과 validation을 구분하였다.
(batch size가 4로 매우 작은 편인데 batch size를 좀만 늘려도 OOM(Out of Memory) 문제가 발생하여서 batch size와 epoch를 모두 줄이게 되었다.)
3. 모델 architecture 정의
먼저 참고한 사이트는 다음과 같다.
https://github.com/meetps/pytorch-semseg/blob/master/ptsemseg/models/unet.py
pytorch-semseg/ptsemseg/models/unet.py at master · meetps/pytorch-semseg
Semantic Segmentation Architectures Implemented in PyTorch - meetps/pytorch-semseg
github.com
https://www.kaggle.com/code/pedroamavizca/working-with-u-net
Working with U-net
Explore and run machine learning code with Kaggle Notebooks | Using data from Cancer Instance Segmentation and Classification 1
www.kaggle.com
이번에 U-Net class를 만들 때 구현하고자 하는 내용은 다음과 같다.
- U-Net 구조
- pixel-wise weight와 cross entropy를 결합한 custom_loss
- mirroring extrapolation
그래서 먼저 U-Net 구조를 만들면
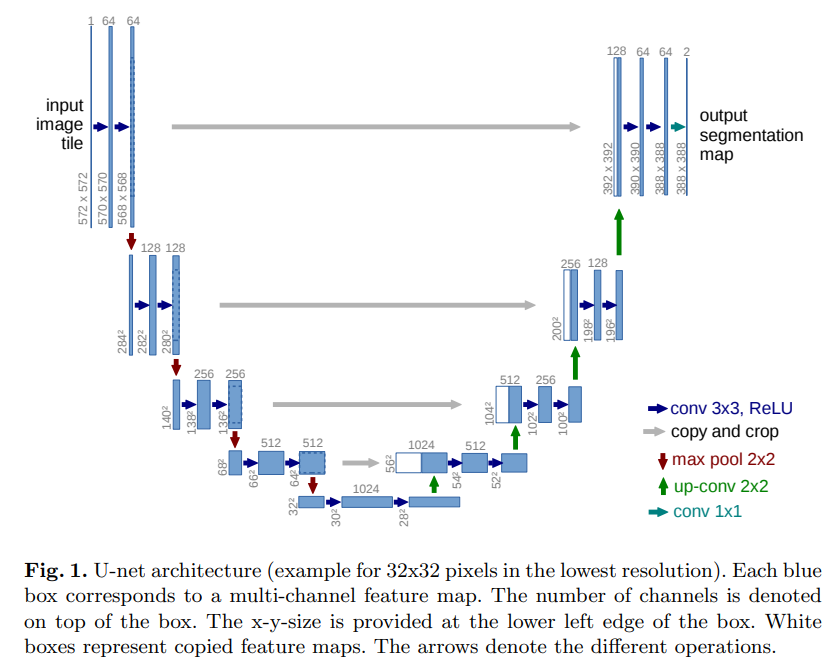
이 구조를 만들어야 하기 때문에 반복적으로 있는 conv 3*3, ReLU(위 그림에서 파란색 2개)를 하나의 class로 정의해보겠다.
class UNetConv2(nn.Module):
def __init__(self, in_channels, out_channels):
super(UNetConv2, self).__init__()
# (입력크기+2*패딩-커널크기)/스트라이드+1
self.conv1 = nn.Sequential(
nn.Conv2d(in_channels, out_channels, kernel_size=3, stride=1, padding=0),
nn.BatchNorm2d(out_channels),
nn.ReLU()
)
self.conv2 = nn.Sequential(
nn.Conv2d(out_channels, out_channels, kernel_size=3, stride=1, padding=0),
nn.BatchNorm2d(out_channels),
nn.ReLU()
)
def forward(self, x):
x = self.conv1(x)
x = self.conv2(x)
return x그 다음 이를 활용하는 전체 U-Net class를 정의하면
class UNet(nn.Module):
def __init__(self, num_classes=6, in_channel=3):
# 부모 클래스 초기화되어 자식 클래스에서도 사용 가능
super(UNet, self).__init__()
self.conv_1 = UNetConv2(in_channel, 64)
self.conv_2 = UNetConv2(64, 128)
self.conv_3 = UNetConv2(128, 256)
self.conv_4 = UNetConv2(256, 512)
self.mid_conv = UNetConv2(512, 1024)
self.conv_5 = UNetConv2(1024, 512)
self.conv_6 = UNetConv2(512, 256)
self.conv_7 = UNetConv2(256, 128)
self.conv_8 = UNetConv2(128, 64)
self.down = nn.MaxPool2d(kernel_size=2, stride=2)
self.up_1 = nn.ConvTranspose2d(1024, 512, kernel_size=2, stride=2)
self.up_2 = nn.ConvTranspose2d(512, 256, kernel_size=2, stride=2)
self.up_3 = nn.ConvTranspose2d(256, 128, kernel_size=2, stride=2)
self.up_4 = nn.ConvTranspose2d(128, 64, kernel_size=2, stride=2)
self.end = nn.Conv2d(64, num_classes, kernel_size=1, stride=1)
def forward(self, x):
padded_x = F.pad(x, (92, 92, 92, 92), mode='reflect')
conv_1 = self.conv_1(padded_x) # output: 252*252
if conv_1.size()[2] % 2 != 0:
conv_1 = F.pad(conv_1, (0, 1, 0, 1))
pool1 = self.down(conv_1) # output: 126*126
conv_2 = self.conv_2(pool1) # output: 122*122
if conv_2.size()[2] % 2 != 0:
conv_2 = F.pad(conv_2, (0, 1, 0, 1))
pool2 = self.down(conv_2) # output: 61*61
conv_3 = self.conv_3(pool2) # output: 57*57
if conv_3.size()[2] % 2 != 0:
conv_3 = F.pad(conv_3, (0, 1, 0, 1))
pool3 = self.down(conv_3) # output: 29*29
conv_4 = self.conv_4(pool3) # output: 25*25
if conv_4.size()[2] % 2 != 0:
conv_4 = F.pad(conv_4, (0, 1, 0, 1))
pool4 = self.down(conv_4) # output: 13*13
mid_conv = self.mid_conv(pool4) # output: 9*9
up_1 = self.up_1(mid_conv)
scale_idx_1 = (conv_4.shape[2] - up_1.shape[2]) // 2
cropped_conv_4 = conv_4[:, :, scale_idx_1:-scale_idx_1, scale_idx_1:-scale_idx_1]
up_1 = torch.cat([up_1, cropped_conv_4], dim=1)
conv_5 = self.conv_5(up_1)
up_2 = self.up_2(conv_5)
scale_idx_2 = (conv_3.shape[2] - up_2.shape[2]) // 2
cropped_conv_3 = conv_3[:, :, scale_idx_2:-scale_idx_2, scale_idx_2:-scale_idx_2]
up_2 = torch.cat([up_2, cropped_conv_3], dim=1)
conv_6 = self.conv_6(up_2)
up_3 = self.up_3(conv_6)
scale_idx_3 = (conv_2.shape[2] - up_3.shape[2]) // 2
cropped_conv_2 = conv_2[:, :, scale_idx_3:-scale_idx_3, scale_idx_3:-scale_idx_3]
up_3 = torch.cat([up_3, cropped_conv_2], dim=1)
conv_7 = self.conv_7(up_3)
up_4 = self.up_4(conv_7)
scale_idx_4 = (conv_1.shape[2] - up_4.shape[2]) // 2
cropped_conv_1 = conv_1[:, :, scale_idx_4:-scale_idx_4, scale_idx_4:-scale_idx_4]
up_4 = torch.cat([up_4, cropped_conv_1], dim=1)
conv_8 = self.conv_8(up_4)
end = self.end(conv_8)
scale_idx_5 = (end.shape[2]-x.shape[2]) // 2
end = end[:, :, scale_idx_5:-scale_idx_5, scale_idx_5:-scale_idx_5]
return end 다음과 같은데 최대한 논문에서 말하는 구조를 따라하기 위해 padding을 0으로 하고 croppping하는 과정을 넣었는데 참고한 사이트에선 구현을 용이하기 위해 padding을 사용하였다. 그리고 논문에서 segmentation이 용이하게 되기 위해서는 max pooling을 하는 input의 size가 짝수여야 했는데 이미 이미지의 크기가 256*256으로 제한되어 있어서 이를 늘리거나 줄이지는 않고 홀수 일때만 padding을 추가하는 방식으로 구현하였다.
torch.summary를 사용해 출력해보면 다음과 같다.
torch.Size([500, 256, 256])
torch.Size([500, 256, 256])
torch.Size([500, 256, 256])
torch.Size([500, 256, 256])
torch.Size([500, 256, 256])
torch.Size([156, 256, 256])
torch.Size([2656, 256, 256])
(2656, 256, 256, 3)
(2656, 256, 256, 6)
torch.Size([16, 3, 256, 256])
----------------------------------------------------------------
Layer (type) Output Shape Param #
================================================================
Conv2d-1 [-1, 64, 438, 438] 1,792
BatchNorm2d-2 [-1, 64, 438, 438] 128
ReLU-3 [-1, 64, 438, 438] 0
Conv2d-4 [-1, 64, 436, 436] 36,928
BatchNorm2d-5 [-1, 64, 436, 436] 128
ReLU-6 [-1, 64, 436, 436] 0
UNetConv2-7 [-1, 64, 436, 436] 0
MaxPool2d-8 [-1, 64, 218, 218] 0
Conv2d-9 [-1, 128, 216, 216] 73,856
BatchNorm2d-10 [-1, 128, 216, 216] 256
ReLU-11 [-1, 128, 216, 216] 0
Conv2d-12 [-1, 128, 214, 214] 147,584
BatchNorm2d-13 [-1, 128, 214, 214] 256
ReLU-14 [-1, 128, 214, 214] 0
UNetConv2-15 [-1, 128, 214, 214] 0
MaxPool2d-16 [-1, 128, 107, 107] 0
Conv2d-17 [-1, 256, 105, 105] 295,168
BatchNorm2d-18 [-1, 256, 105, 105] 512
ReLU-19 [-1, 256, 105, 105] 0
Conv2d-20 [-1, 256, 103, 103] 590,080
BatchNorm2d-21 [-1, 256, 103, 103] 512
ReLU-22 [-1, 256, 103, 103] 0
UNetConv2-23 [-1, 256, 103, 103] 0
MaxPool2d-24 [-1, 256, 52, 52] 0
Conv2d-25 [-1, 512, 50, 50] 1,180,160
BatchNorm2d-26 [-1, 512, 50, 50] 1,024
ReLU-27 [-1, 512, 50, 50] 0
Conv2d-28 [-1, 512, 48, 48] 2,359,808
BatchNorm2d-29 [-1, 512, 48, 48] 1,024
ReLU-30 [-1, 512, 48, 48] 0
UNetConv2-31 [-1, 512, 48, 48] 0
MaxPool2d-32 [-1, 512, 24, 24] 0
Conv2d-33 [-1, 1024, 22, 22] 4,719,616
BatchNorm2d-34 [-1, 1024, 22, 22] 2,048
ReLU-35 [-1, 1024, 22, 22] 0
Conv2d-36 [-1, 1024, 20, 20] 9,438,208
BatchNorm2d-37 [-1, 1024, 20, 20] 2,048
ReLU-38 [-1, 1024, 20, 20] 0
UNetConv2-39 [-1, 1024, 20, 20] 0
ConvTranspose2d-40 [-1, 512, 40, 40] 2,097,664
Conv2d-41 [-1, 512, 38, 38] 4,719,104
BatchNorm2d-42 [-1, 512, 38, 38] 1,024
ReLU-43 [-1, 512, 38, 38] 0
Conv2d-44 [-1, 512, 36, 36] 2,359,808
BatchNorm2d-45 [-1, 512, 36, 36] 1,024
ReLU-46 [-1, 512, 36, 36] 0
UNetConv2-47 [-1, 512, 36, 36] 0
ConvTranspose2d-48 [-1, 256, 72, 72] 524,544
Conv2d-49 [-1, 256, 70, 70] 1,179,904
BatchNorm2d-50 [-1, 256, 70, 70] 512
ReLU-51 [-1, 256, 70, 70] 0
Conv2d-52 [-1, 256, 68, 68] 590,080
BatchNorm2d-53 [-1, 256, 68, 68] 512
ReLU-54 [-1, 256, 68, 68] 0
UNetConv2-55 [-1, 256, 68, 68] 0
ConvTranspose2d-56 [-1, 128, 136, 136] 131,200
Conv2d-57 [-1, 128, 134, 134] 295,040
BatchNorm2d-58 [-1, 128, 134, 134] 256
ReLU-59 [-1, 128, 134, 134] 0
Conv2d-60 [-1, 128, 132, 132] 147,584
BatchNorm2d-61 [-1, 128, 132, 132] 256
ReLU-62 [-1, 128, 132, 132] 0
UNetConv2-63 [-1, 128, 132, 132] 0
ConvTranspose2d-64 [-1, 64, 264, 264] 32,832
Conv2d-65 [-1, 64, 262, 262] 73,792
BatchNorm2d-66 [-1, 64, 262, 262] 128
ReLU-67 [-1, 64, 262, 262] 0
Conv2d-68 [-1, 64, 260, 260] 36,928
BatchNorm2d-69 [-1, 64, 260, 260] 128
ReLU-70 [-1, 64, 260, 260] 0
UNetConv2-71 [-1, 64, 260, 260] 0
Conv2d-72 [-1, 6, 260, 260] 390
================================================================
Total params: 31,043,846
Trainable params: 31,043,846
Non-trainable params: 0
----------------------------------------------------------------
Input size (MB): 0.75
Forward/backward pass size (MB): 1773.24
Params size (MB): 118.42
Estimated Total Size (MB): 1892.42
----------------------------------------------------------------4. 손실함수와 optimizer 정의
우선 model과 optimizer는 쉽게 정의했는데
model = UNet().to(device)
optimizer = torch.optim.Adam(model.parameters(), lr=0.01)일반적인 loss fucntion을 사용하는 것이 아닌 각 pixel에 weight를 부여하고 softmax함수와 cross entropy loss를 결합한 loss(논문에 잘 소개되어있다.)를 사용해보기 위해 함수들을 정의해보았다.
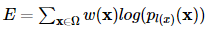
def custom_loss(outputs, labels, weights):
# softmax 계산
softmax_outputs = F.softmax(outputs, dim=1)
# CPU로 이동
labels = labels.cpu()
weights = weights.cpu()
softmax_outputs = softmax_outputs.cpu()
# 0이 아닌 위치를 찾기 위한 마스크 생성
non_zero_mask = labels != 0
# 마스크를 사용하여 필요한 값 선택 및 계산
selected_weights = weights.unsqueeze(1).expand_as(labels)[non_zero_mask]
selected_softmax_outputs = softmax_outputs[non_zero_mask]
# 손실 계산
running_loss = (-1) * selected_weights * torch.log(selected_softmax_outputs)
running_loss = running_loss.sum()
running_loss /= labels.shape[0] * labels.shape[2] * labels.shape[3]
return running_loss.to(outputs.device) 먼저 outputs에서 softmax를 적용하고(채널이 class별 출력을 위하니 dim=1로 softmax를 계산한다.) labels에서 0이 아닌 위치를 나타내는 non_zero_mask를 정의하였다. weights의 차원은 (batch_size, height, width)이고 non_zero_mask의 차원은 (batch_size, num_classes, height, width)에서 selected_weights를 구하기 위해선 weights에서 두번째 차원을 추가하고 labels의 num_classes만큼 복사한다음 선택을 한다. 그렇게 계산한 selected_weights의 차원은 non_zero의 갯수가 N이라 하면 (N,)이다. 그리고 필는 loss function을 최소화하기 위해 -1을 붙였고 pixel-wise loss때문에 평균을 내기 위해 batch_size와 height, width로 나누어줬다.
그럼 weight는 어떻게 계산할까? 논문에서는
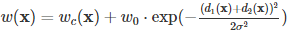
이렇게 class의 frequency를 반영한 1차 weight에 세포와의 거리를 반영하는 추가적인 weight로 계산하였는데 구현이 엄청 어려운 것이지만 시간이 매우 오래 걸릴 것이라 판단하여 비슷한 아이디어로 정의하였다.
import torch
def find_others(labels, i, j, k, b, d):
left = max(i - d, 0)
right = min(i + d, 255) # 256이 아니라 255까지
up = max(j - d, 0)
down = min(j + d, 255) # 256이 아니라 255까지
instance = labels[b, k, i, j]
region = labels[b, k, left:right+1, up:down+1]
other_classes = (region == 0).sum().item()
other_instances = ((region != 0) & (region != instance)).sum().item()
return other_classes, other_instances
def calculate_weights(masks):
device = masks.device
batch_size, num_classes, height, width = masks.shape
weights = torch.zeros((batch_size, height, width), device=device)
non_zero_counts = (masks != 0).sum(dim=(2, 3))
for b in range(batch_size):
non_zero_ratio = non_zero_counts[b].float() / non_zero_counts[b].sum(dim=0, keepdim=True).float()
exp_non_zero_ratio = torch.exp(-non_zero_ratio)
for k in range(num_classes):
mask_k = masks[b, k]
non_zero_mask = mask_k != 0
weights[b][non_zero_mask] = exp_non_zero_ratio[k]
for i in range(2, height, 5):
for j in range(2, width, 5):
if non_zero_mask[i, j]:
other_classes, other_instances = find_others(masks, i, j, k, b, 2)
weights[b, i-2:i+3, j-2:j+3] *= (1.02)**other_classes
weights[b, i-2:i+3, j-2:j+3] *= (1.05)**other_instances
return weights 기본적인 아이디어는 먼저 class의 frequency의 비율에 exp(-x)를 적용하고 해당 pixel을 가운데로 하는 25개의 pixel에 같은 class지만 다른 instance인 pixel의 개수(a)와 다른 class인 pxiel의 개수(b)에 따라 가중치를 각각 1.05**(a), 1.02**(b) 배 해주는 방식이다. 총 2656개의 데이터의 256*256 pixels는 1억개가 넘어서(174,063,616) 다섯 pixels씩 넘어가면서 계산하도록 설계하였다.
matplotlib의 pyplot을 사용하여 하나의 weight를 시각해보면 다음과 같다.
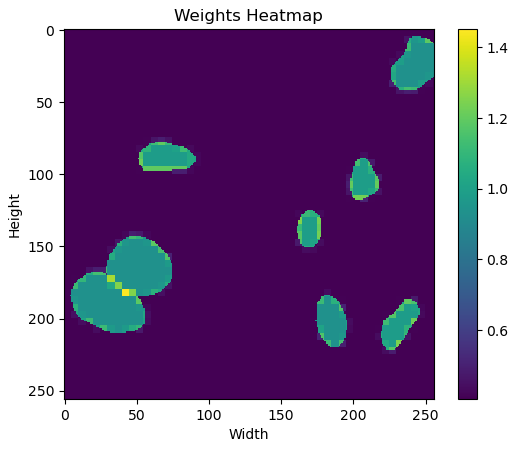
경계에서, 특히 다른 instance의 경계에서 가장 높은 weight를 갖도록 설계된 것을 알 수있다.
5. Train & Validation
num_epochs = 50 # Number of epochs
batch_size = 4
val_idx_start = len(train_loader.dataset)
for epoch in range(num_epochs):
print(f'Epoch {epoch+1}/{num_epochs}')
# Each epoch has a training and validation phase
for phase in ['train', 'val']:
if phase == 'train':
model.train() # Set model to training mode
dataloader = train_loader
else:
model.eval() # Set model to evaluate mode
dataloader = val_loader
running_loss = 0.0
# Iterate over data with tqdm for the progress bar
progress_bar = tqdm(enumerate(dataloader), total=len(dataloader), desc=f"{phase.capitalize()} Phase")
for batch_idx, (inputs, labels) in progress_bar:
inputs, labels = inputs.to(device), labels.to(device)
# Zero the parameter gradients
optimizer.zero_grad()
# Forward
with torch.set_grad_enabled(phase == 'train'):
outputs = model(inputs)
if phase == 'train':
batch_weights = weights[batch_idx * batch_size : (batch_idx + 1) * batch_size]
else:
batch_weights = weights[val_idx_start + batch_idx * batch_size : val_idx_start + (batch_idx + 1) * batch_size]
loss = custom_loss(outputs, labels, batch_weights)
if phase == 'train':
loss.backward()
optimizer.step()
# Statistics
running_loss += loss.item() * inputs.size(0)
epoch_loss = running_loss / len(dataloader.dataset)
# Update the progress bar with the current loss value
progress_bar.set_postfix({'loss': f'{loss.item():.4f}'})
if phase == 'val':
print(f'{phase.capitalize()} Loss: {epoch_loss:.4f}')
print()GPU 메모리와 사용량의 한계로 epoch를 50으로 설정하였고 위에서 계산한 weight를 batch에 맞게 가져오도록 설계하였다. 훈련은 총 3시간 정도 걸렸다.
결과를 시각화해보면
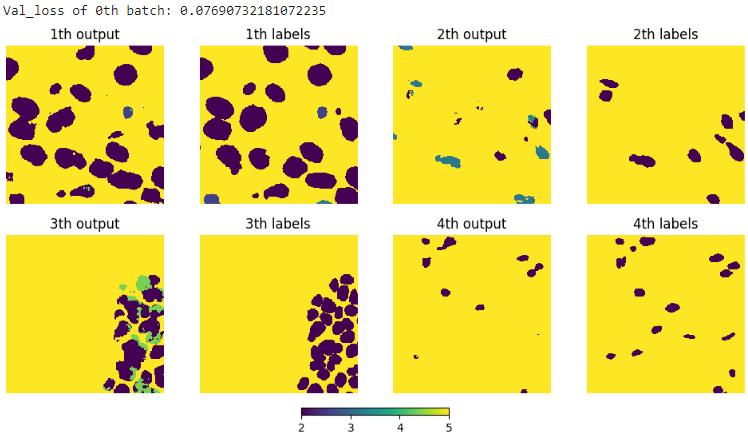
이렇게 생각보다 output이 labels를 잘 분류하는 것을 알 수 있다. 그런데 필자의 최종 목표는 instance segmentation이기 때문에 watershed 알고리즘을 적용하였다. 아래 그림은 바로 위 그림의 첫번째 output에 적용한 결과인데
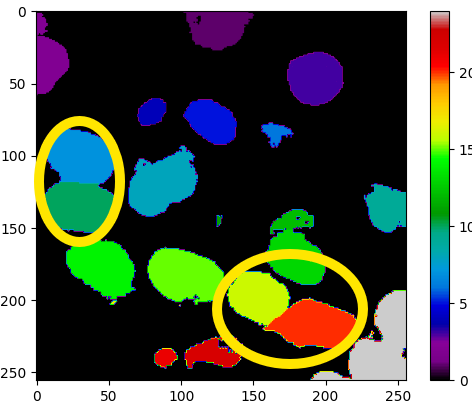
노란색 타원으로 표시한 부분처럼 touching instances들도 구분한 것을 알 수 있다.
+ watershed 알고리즘
객체를 분할하는데 사용되는 기법으로 지형학적 모델을 사용하여 이미지의 픽셀을 분할하는기 때문에 outputs의 각 클래스별 출력이 높이로 간주된다. 이 알고리즘은 물이 채워질 때 계곡을 따라 경계가 형성된다는 개념에서 유래됐다. 알고리즘의 단계로는
1. 전처리 - threshold 보다 낮은 값들은 제거한다.
2. 거리 변환 - 객체 내부의 각 픽셀이 가장 가까운 배경 픽셀로부터 얼마나 떨어져 있는지 계산한다. 여기서 거리는 Manhattan distance를 의미한다.
3. 마커 생성 - 지역 극대값을 찾아 마커로 설정한다. 마커는 객체의 중심을 나타낸다.
4. watershed 변환 - 마커에서 시작하여 물을 채워 나가, 서로 다른 마커에서 채워진 물이 만나는 지점에서 경계가 형성된다.
가 있고 그 결과 위 그림처럼 instance segmentation이 수행된다.
6. 마무리
위 전체 코드를 구현한 것은 깃허브에 올려두었다.
https://github.com/ParkSeokwoo/U-Net-cancer-instance-segmentation-
GitHub - ParkSeokwoo/U-Net-cancer-instance-segmentation-
Contribute to ParkSeokwoo/U-Net-cancer-instance-segmentation- development by creating an account on GitHub.
github.com
Watershed 알고리즘을 진작에 알았다면 loss를 설계하는 과정이 더 논문에 가까워졌을 것 같지만 이 정도만으로 만족하고 넘어가려한다. 그리고 이건 위 모델하고는 상관없는데 생각보다 kaggle notebook이 괜찮은거 같다. Colab pro를 결제해도 다 쓰는데 1주일 밖에 걸리지 않는데 kaggle notebook은 전화번호가 있는데로 쓸 수 있고 1주일마다 다시 30시간을 사용할 수 있기 때문에 앞으로 대부분의 작업을 kaggle에서 사용할 것 같다. 다음 포스트는 동아리 사람들과 diffusion 모델 스터디를 하게 되어서 스터디 준비용 포스트를 올릴 것 같다. 바이~
'DL > Image Segmentation' 카테고리의 다른 글
| [Paper Reivew] U-Net: Convolutional Networks for Biomedical Image Segmentation (0) | 2024.05.27 |
|---|---|
| FCN을 구현해보자!(sub task: cloth segmentation) (0) | 2024.05.20 |
| [Paper Review] Fully Convolutional Netowrks for Semantic Segmentation (0) | 2024.05.06 |